Lirola-Andreu, Laura¹*; Ávila-Jiménez, Ángel Francisco¹; Fernández-Mariscal, Marta Andrea¹;
Reinoso-Espín, Ángela¹; Martínez-Martínez, Silvia¹
1 Facultad de Medicina, Universidad de Granada (UGR)
* Autor de correspondencia: lirolaandreulaura@gmail.com
Resumen
En la actualidad, la resistencia a los antibióticos es un problema de gran impacto en la salud a nivel mundial. Los datos de grandes organizaciones mundiales como la Organización Mundial de la Salud apuntan a que se trata de un peligro real que requiere de inmediata actuación. La resistencia a los antibióticos es consecuencia directa del uso inapropiado de dichos medicamentos. Los antibióticos fueron concebidos para acabar con las infecciones bacterianas mediante distintos mecanismos. No obstante, debido a su abuso, están provocando una presión selectiva sobre las poblaciones bacterianas, favoreciendo la “supervivencia de los más aptos”. Por otro lado, las bacterias son capaces de generar resistencia a dichos fármacos, ya sea a través de mutaciones o por la adquisición de nuevo material genético. Los mecanismos de resistencia son múltiples y muy diversos. Sin embargo, por su especial protagonismo en los últimos años destaca un proceso de inhibición enzimática englobado dentro del grupo de las 𝛽-lactamasas, las carbapenemasas. Existen muchos tipos de carbapenemasas, cada una con sus características particulares, y que son producidas por un amplio espectro de bacterias. En particular, se hace especial alusión a tres tipos de carbapenemasas de gran relevancia en los últimos años: las KPC (Klebsiella Pneumoniae Carbapenemases), las NDM (New Delhi Metallo-𝛽-lactamases) y las similares a OXA-48. Por último, ante la aplastante evidencia, los esfuerzos por buscar soluciones y alternativas no cesan. En España se están llevando a cabo múltiples investigaciones y proyectos destinados a este propósito. Por ejemplo, se presentan el Plan Nacional frente a la Resistencia a los Antibióticos o, ya en el ámbito de la investigación, la participación del Centro Nacional de Biotecnología.
Palabras clave: resistencia bacteriana, mecanismos, antibióticos, carbapenemasas, investigaciones.
Keywords: antimicrobial resistance, mechanisms, antibiotics, carbapenemases, research.
1. Introducción
Una bacteria multirresistente es aquella que desarrolla resistencia a más de un antibiótico. En la actualidad, la resistencia bacteriana supone una de las mayores preocupaciones de las organizaciones de salud de todo el mundo. Es un problema que ya afecta a la práctica clínica: cada vez es más común en centros de salud y hospitales; en ocasiones, conlleva el fracaso y la complicación de los tratamientos, debido al desarrollo de infecciones y cuadros sépticos que no pueden ser solucionados con antibióticos. En pacientes, especialmente en los más críticos, esto genera graves consecuencias.
La Organización Mundial de la Salud (OMS) publicó en 2017 la lista de “patógenos prioritarios” (1), resaltando la necesidad de desarrollar estrategias que ayuden a combatir una problemática que, dentro de unos años, podría convertirse en una pandemia de magnitudes inimaginables. En dicha lista y, entre los de prioridad más crítica se destacan algunos patógenos resistentes a antibióticos carbapenémicos. En prioridad elevada se encuentran Staphylococcus aureus resistente a la meticilina (SARM o, en inglés, MRSA) y Staphylococcus aureus resistente a la vancomicina (SARV), entre otros.
Se plantea entonces el problema de la adquisición de resistencia a los antibióticos. Algunos componentes de resistencia aparecen de forma natural. Sin embargo, es el uso inadecuado de antibióticos, a nivel sanitario y en la industria alimentaria (especialmente la ganadera), el que está propiciando la aparición de cepas cada vez más resistentes. Según el Centro Europeo de Prevención y Control de Enfermedades (ECDC): “en 2018, ciento cincuenta y cuatro países acordaron aumentar la vigilancia y el uso y abuso del consumo de antibióticos en humanos y ganado” (2).
En contrapartida, en los últimos años el desarrollo de nuevos antibióticos ha sido escaso o nulo y se están agotando el número de antibióticos eficaces. Es más, desde la “década de oro” del desarrollo de antibióticos iniciada con el descubrimiento de la penicilina, sólo se han aprobado tres clases de antibióticos contra bacterias Gram Positivas: oxazolidinona, daptomicina y fidaxomicina (3). Como consecuencia, la adquisición de resistencia por parte de las bacterias sucede a una velocidad mayor que el desarrollo de contraataques.
Esta revisión tiene como objetivo dar a conocer la importancia del fenómeno de la resistencia a antibióticos, alentando a comprender mejor este asunto, su relevancia y su gravedad. Asimismo, se pretende explicar brevemente en qué consisten los mecanismos que dotan a las bacterias de dicha resistencia, profundizando en uno de creciente preocupación, como lo es el mecanismo enzimático de las carbapenemasas. Finalmente, dar una visión general sobre las últimas investigaciones y proyectos puestos en marcha, fundamentalmente en España, para hacer frente a este problema.
2. Antibióticos y mecanismos de resistencia bacteriana
Para comprender los mecanismos de resistencia bacteriana resulta esencial conocer primero cómo funcionan los antibióticos. El término antibiótico procede de la palabra griega antibiosis (αντίβιος) que significa “contra la vida”. Se refiere, en términos generales, a aquella sustancia natural o sintética que mata o inhibe las actividades metabólicas de bacterias y otros microorganismos (4).
La mayor parte de los antibióticos pueden ser categorizados en cuatro grandes grupos según su principal mecanismo de acción: inhibición de la síntesis de la pared bacteriana, impedimento de la síntesis proteica, bloqueo de la síntesis de ácidos nucleicos e inhibición de la ruta metabólica (tabla 1) (5,6).

Dentro de la primera categoría, los antibióticos β-lactámicos, entre los que destacan los carbapenémicos, representan el grupo más amplio. Los β-lactámicos actúan como enzimas hidrolíticas que irrumpen las fases finales de la síntesis de la pared bacteriana, lo que resulta en la pérdida de la integridad anatomofisiológica de la bacteria y la destrucción del microorganismo. Para cumplir su función, los β-lactámicos se unen covalentemente a las penicillin-binding-protein (PBPs), también llamadas genéricamente como “transpeptidasas”, las cuales son necesarias para el ensamblaje de los peptidoglicanos de la pared celular (7,8).
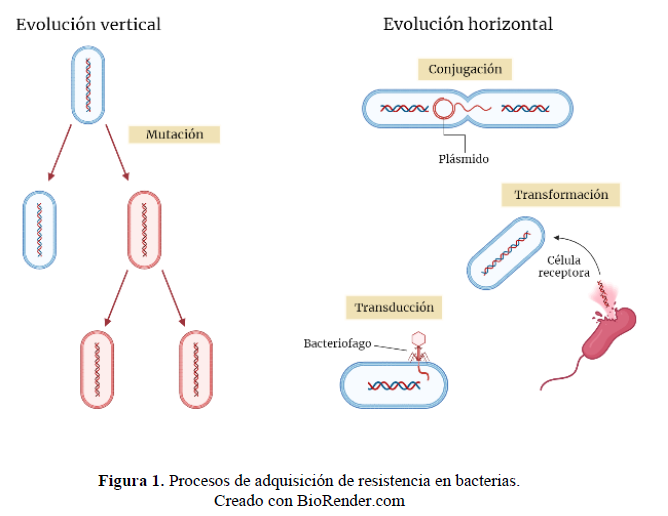
No obstante, las bacterias son capaces de manifestar resistencia frente a los antibióticos de dos formas: innata y/o adquirida. Algunas especies de bacterias presentan características estructurales y funcionales únicas que las dotan de una “resistencia innata” a ciertos tipos de agentes antimicrobianos (9). Sin embargo, es la “resistencia adquirida” por mutaciones o adopción de material genético la que resulta más preocupante. Las bacterias pueden adquirir resistencia mediante mutaciones (puntuales o estructurales). Dichas mutaciones son sometidas a una presión selectiva por los antimicrobianos. La presión selectiva favorece la supervivencia de las cepas más resistentes y da lugar a una “evolución vertical” (3,4). Asimismo, las bacterias pueden desarrollar resistencia a través de la adquisición de nuevo material genético proveniente de otros microorganismos resistentes. Esto se conoce como “evolución horizontal” y se puede dar por 3 procesos: transformación, conjugación y transducción (figura 1) (4)
Una vez adquirida la nueva información, la resistencia bacteriana puede aparecer mediante múltiples mecanismos. Se diferencian cuatro mecanismos principales:
- Alteraciones en el lugar de acción. Consiste en la modificación del lugar de unión del antibiótico, ya sea a través de la alteración de sus precursores, de forma directa sobre la enzima diana o protegiendo los sitios diana (7).
- Disminución de la permeabilidad. Las porinas son la principal vía de entrada de los antibióticos hidrofílicos. Las mutaciones que produzcan la pérdida, modificación o reducción de la expresión de porinas; determinarán una mayor resistencia a los antibióticos que precisan de entrar en el interior celular para inutilizar a la bacteria (9,10).
- Bomba de expulsión. Las bombas de expulsión son sistemas complejos dependientes de energía (ATP o fuerza protón motriz) presentes en la membrana citoplasmática bacteriana. Estos sistemas normalmente son utilizados para bombear fuera de la célula moléculas tóxicas procedentes del metabolismo celular. Algunas bacterias, sin embargo, pueden emplearlos para expulsar al antibiótico (4,11).
- Inactivación o modificación enzimática. Consiste en la adición, por parte de enzimas bacterianas, de grupos fosfato, acetilo o adenilo en sitios específicos del antibiótico; modificándolo químicamente e inactivándolo, evitando así que se una a su diana (7).
2.1. Las β-lactamasas, una resistencia frente a los β-lactámicos
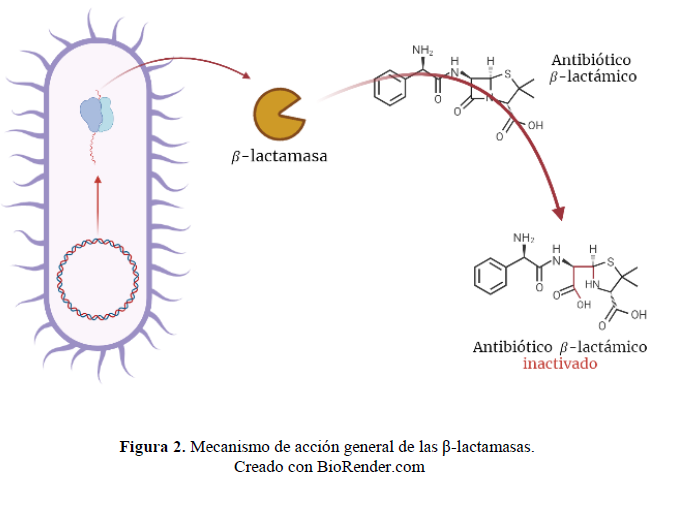
La resistencia a los antibióticos β-lactámicos tiene lugar a través de diversos mecanismos: la alteración de la permeabilidad o expulsión activa, la alteración de la estructura de los PBPs, o, principalmente, mediante la producción de β-lactamasas que rompen los anillos β-lactámicos (figura 2) (12).
Las β-lactamasas pueden clasificarse en cuatro grupos (A, B, C y D) en función de su estructura molecular. De igual manera, pueden ser clasificadas en dos categorías en función de sus propiedades funcionales: las β-lactamasas de amplio espectro y las Carbapenemasas (11). Estas últimas, por su importancia, se desarrollarán en el siguiente apartado.
3. Carbapenemasas
Las carbapenemasas integran a un amplio grupo de enzimas pertenecientes a las β-lactamasas. Según la clasificación Ambler (13), que atiende a sus características estructurales y funcionales, se distinguen:
- Clase A (Serin-β-lactamasas): presentan un residuo de serina en su centro activo. Incluye a las Klebsiella Pneumoniae Carbapenemases (KPC), NmcA, IMI, Serratia Marcescens Enzyme (SME), Guiana Extended-Spectrum (GES) y Serratia Fonticola Carbapenemases (SFC) entre otras (14,15). Algunas se hallan codificadas en cromosomas y otras en plásmidos. Tienen capacidad de hidrolizar carbapenémicos y son parcialmente inhibidas por el ácido clavulánico (16–19).
- Clase B (Metalo-β-lactamasas (MBLs)): presentan un ion metálico como cofactor enzimático. Dentro de este grupo se encuentran las New Delhi metallo-β-lactamases (NDM) y las IMP (15). Tienen la capacidad de hidrolizar todas las β-lactamasas a excepción del aztreonam. Son inhibidas por el ácido etilendiaminotetraacético (EDTA), pero no por el ácido clavulánico. Las bacterias que las expresan causan predominantemente infecciones nosocomiales y presentan multirresistencia (16–19).
- Clase D (Oxaciclinasas): tienen la capacidad añadida de hidrolizar oxaciclina y cloxacilina. Dentro de este grupo destacan las OXA-48-like enzymes (15). En concreto, OXA-48 y OXA-181 son particularmente interesantes por su escasa capacidad para hidrolizar carbapenémicos y cefalosporinas. No obstante, ni el ácido clavulánico ni el EDTA han demostrado eficacia en disminuir su mecanismo de acción (14–17).
Los genes relacionados con la producción de carbapenemasas permiten obtener una forma de resistencia estable, que puede ser propagada por expansión clonal y/o por transferencia genética horizontal a través de plásmidos (17). Las bacterias productoras de carbapenemasas, por su facilidad de expansión y su elevada mortalidad, requieren de una supervisión exhaustiva para evitar su proliferación. Además, los procedimientos necesarios para el reconocimiento de las carbapenemasas no están implementados en la mayoría de laboratorios. La capacidad de distinguir entre microorganismos productores y no productores de carbapenemasas, así como el hecho de poder distinguir entre subgrupos podría ser una poderosa herramienta tanto para el diagnóstico como para el tratamiento.
En la actualidad, se conocen gran multitud de microorganismos que tienen la capacidad de producir este tipo de enzimas, algunos de los cuales se exponen a continuación.
3.1. Bacterias productoras de KPC
KPC es el subtipo de carbapenemasa de clase A más extendido a nivel global entre las Enterobacteriaceae spp. (17). Actualmente hay descritas hasta 22 variantes, que forman parte de las bacterias Gram negativas. La primera bacteria descrita que implementaba estas enzimas como mecanismo de resistencia fue Klebsiella pneumoniae, descubierta en Carolina del Norte (EEUU) en 1996. Poco después, a mediados del año 2000, K. pneumoniae se extendió a diversos países como Israel, Grecia y Colombia, entre otros. La técnica más utilizada para la identificación y seguimiento de los brotes de esta bacteria es la tipificación multilocus de secuencias (MLST). La MLST es un procedimiento en el que se caracterizan muestras de especies microbianas por secuenciación de ADN, asignando un número de alelo a cada secuencia única y comparándola con las existentes en las bases de datos.
Seguidamente, se analizan algunos aspectos epidemiológicos de esta bacteria en algunos países:
- Este país ha mostrado una de las tasas más elevadas de resistencia a los carbapenémicos en los últimos años. El Sistema Griego para la Supervisión de la Resistencia Bacteriana descubrió un aumento en la prevalencia de la resistencia a los carbapenémicos de menos del 1% en el año 2001 hasta un 30% en 2008 en hospitales (20). Particularmente, los brotes de K. pneumoniae productoras de blaKPC-2 fueron por primera vez notificados en el año 2007. En 2008, un estudio que analizó 21 hospitales encontró casos en 18 de ellos (21). Estudios más actuales han demostrado que la variante dominante de K. pneumoniae productora de KPC es ST258 (22).
- Estados Unidos. Como se ha mencionado antes, la primera bacteria productora de KCP se descubrió en Carolina del Norte en 1996. En 2001 se produjo un gran aumento de casos de pacientes infectados con bacterias productoras de KPC con foco en Nueva York (20). Posteriormente, en 2006, la Recogida de Información de la Prueba de Susceptibilidad Anual de Meropenem (en inglés, MYSTIC) detectó 57 nuevos brotes (23). Una encuesta a nivel nacional realizada por el Programa de Vigilancia Antimicrobiana (SENTRY) entre 2007 y 2009 estudió 42 centros médicos y encontró una prevalencia del 5.5% en bacterias productoras de blaKPC (24). En la población infantil también se ha detectado cierto incremento en la prevalencia de bacterias resistentes a carbapenémicos, aunque no hay mucha información accesible (25). Un estudio realizado algunos años atrás recopiló datos de alrededor de 300 laboratorios que participaban en la Surveillance Network–USA database, y detectó un aumento durante el desarrollo del mismo (enero de 1999 a julio de 2012) del 0.47% de la prevalencia de Enterobacteriaceae spp. Resistentes a Carbapenémicos (22,26–28).
3.2. Bacterias productoras de NDM
Las NDM son un subgrupo de carbapenemasas clase B, del cual existen 24 variantes descritas en más de 60 especies pertenecientes a 11 familias bacterianas. Se caracterizan por tener la capacidad de hidrolizar la mayoría de β-lactamasas, pero no a los monobactámicos. Su actuación no puede ser contrarrestada por inhibidores clínicos convencionales, tales como el avibactam o el ácido clavulánico (29).
Inicialmente, NDM-1 fue detectada en un paciente sueco infectado con K. pneumoniae en Nueva Delhi. Posteriormente, se han encontrado cepas productoras de NDM en varias especies de Enterobacteriaceae spp., Acinetobacter spp. y Pseudomonas spp., y en todo el mundo. Las bacterias productoras de este tipo de carbapenemasa causan gran variedad de infecciones con índices de mortalidad muy elevados (29,30).
Con respecto a su estructura, destacar que se compone de 270 aminoácidos, con 2 iones zinc en el centro activo, donde se hidroliza a los β-lactámicos. En cuanto a su estructura secundaria, presenta 9 α-hélices, 17 láminas β y tres giros (27).
Aunque se están realizando múltiples esfuerzos para contener esta amenaza, las bacterias productoras de NDM siguen expandiéndose y resultan una amenaza para la salud pública a nivel global (31). No hay ningún tratamiento contra las NDM aprobado para uso clínico todavía, pero se están investigando múltiples agentes.
Las principales razones por las que es necesario prestar más atención a este tipo de microorganismos son múltiples:
- Las investigaciones relacionadas con la epidemiología de bacterias productoras de NDM son todavía escasas.
- Todavía no se conoce el ritmo en el que este tipo de bacterias se está propagando.
- Fuera de las instalaciones sanitarias no está clara la expansión de las cepas productoras de NDM.
- No se ha establecido un tratamiento óptimo para este tipo de infecciones (29).
3.3. Bacterias productoras de OXA-48-like enzymes
OXA-48-like enzymes es uno de los grupos de enzimas más comunes en las Enterobacteriaceae spp. Resistentes a Carbapenémicos. Es probable que el progenitor probable de OXA-48 perteneciera a Shewanella spp., una bacteria transmitida por agua (16). Existen más de 15 variantes, pero OXA-48 es la más extendida. La rápida expansión de estos microorganismos se debe principalmente a la posibilidad de transmitir el plásmido que codifica el gen blaOXA-48, localizado en un transposón, facilitando su distribución a otras bacterias (14).
En años recientes, OXA-48 se ha convertido en la carbapenemasa más común en varias regiones geográficas (16,32). El primer contacto con esta enzima se dio en Turquía en el año 2001, en una infección urinaria. Posteriormente se identificaron numerosos brotes nosocomiales en este mismo país. En Europa el primer caso se produjo en Bélgica, en 2008. En África ocurrió en Túnez, sobre el año 2010. En Asia se detectó originariamente en Kuwait, en 2012. En Norte América, en Estados Unidos, en 2013; mientras que en Sudamérica ocurrió en Brasil, en 2014. En total, más de 50 países han reportado brotes de bacterias productoras de OXA-48 (14). En España, el primer caso tuvo lugar en 2013 (16,33).
El problema se hace más complejo, debido a que este tipo de bacteria integra, aparte de este mecanismo de resistencia, otros como la modificación de receptores o presencia de bombas de expulsión. También es importante destacar que la frecuencia de infección de estas bacterias a nivel mundial está en aumento. En España es incluso mayor que la media europea (16,18,33).
4. Investigaciones, actualidad y situación en España
La resistencia bacteriana es un problema a escala mundial. Según datos de la AEMPS (Agencia Española de Medicamentos y Productos Sanitarios) y del PRAN (Plan Nacional frente a la Resistencia a los Antibióticos), las bacterias multirresistentes provocan alrededor de 3000 muertes al año en España, 33000 muertes al año en Europa y 700000 muertes al año en todo el mundo. Además, la resistencia bacteriana genera un gasto sanitario adicional de unos 1500 millones de euros (34,35).
No obstante, los esfuerzos por superar esta situación siguen adelante. En España, destaca el PRAN, cuyo objetivo es reducir el riesgo de resistencia a los antibióticos y el impacto de este problema sobre la salud de las personas y los animales, preservando de manera sostenible la eficacia de los antibióticos existentes (33). Al mismo tiempo, trata de concienciar y formar tanto a profesionales sanitarios como a toda la población. En el PRAN participan una inmensa cantidad de ministerios, sociedades científicas y expertos, entre otros. En marzo de 2022 se anunciaban los ganadores de la III edición de los premios del PRAN, dando reconocimiento a proyectos que luchan contra la situación (34–36).
Por otro lado, la Asociación Española de Pediatría – Sociedad Española de Infectología Pediátrica (AEP-SEIP) hace su aportación y se posiciona sobre el tratamiento de las infecciones por bacterias multirresistentes, revisando la epidemiología y el tratamiento, siguiendo la mejor evidencia disponible (37).
Finalmente, entrando en el ámbito de la investigación, sobresale el Centro Nacional de Biotecnología (CNB) del CSIC, en Madrid. El CNB cuenta con un departamento de Biotecnología Microbiana, en el que trabajan en la resistencia a los antibióticos (mecanismos de resistencia, su adquisición y efectos), entre otras cosas (38).
Un grupo del CNB investiga con microdominios de membrana, como balsas lipídicas en la membrana de las bacterias, las cuales son capaces de modificar para lograr resultados útiles en muchos campos, por ejemplo, favoreciendo la producción de antibióticos. Este equipo logró inhibir la resistencia a los antibióticos de SARM mediante el desensamblaje de proteínas (PBP2a, responsable de la resistencia a la penicilina) de estos microdominios de membrana, desactivando la resistencia y logrando que vuelvan a ser susceptibles al tratamiento (López D. y colaboradores) (39).
Otro grupo del CNB ha estudiado el impacto, en la resistencia, de ambientes libres de antibiótico o con concentraciones subletales. Basándose, por un lado, en la compensación del coste fisiológico asociado que puede ocasionar una inhibición de la proliferación o una disminución de la resistencia. Por otro lado, en la sensibilidad colateral, que la hace más susceptible a un segundo antibiótico. Los investigadores proponen realizar tratamientos interrumpiendo el uso, para poder volver a utilizarlo eficazmente, y/o combinarlo con otros antibióticos ya disponibles a continuación del primero, pudiendo volver al mismo. Identifican así una posible estrategia terapéutica, publicando este estudio en marzo de 2022 (40).
La resistencia bacteriana supone un problema de grandes dimensiones. La literatura al respecto es abundante y, en todo el mundo, se han puesto en marcha investigaciones para encontrar una manera de superar o contrarrestar dicha resistencia. Algunos de esos proyectos se han expuesto ya en este artículo. Además, cabe mencionar que existen otras alternativas en estudio, entre ellas:
– Los mecanismos de sensibilidad colateral, basados en la sensibilización hacia un antibiótico derivada de la resistencia a otro (41).
– El uso de nanopartículas para albergar anticuerpos que mejoren su efectividad frente a los microorganismos multirresistentes (3).
– El uso de fosfomicina, antibiótico de amplio espectro muy eficaz contra muchas bacterias resistentes (42).
Adicionalmente, el Estudio de Prevalencia de las Infecciones Nosocomiales en España (EPINE) publica todos los años un estudio nacional sobre la evolución de la prevalencia de microorganismos multirresistentes a nivel hospitalario (43).
5. Limitaciones del estudio
Resulta extremadamente complicado abarcar en un único trabajo un tema tan amplio como es la resistencia bacteriana, ya que afecta a numerosos y muy diversos campos. Nuestro objetivo era mostrar una visión general de un problema tan complejo como es la resistencia a los antibióticos. La búsqueda de información para la elaboración de este trabajo, llevada a cabo fundamentalmente a través de la base de datos PubMed, ha sido intensa, pero en absoluto cerrada.
6. Conclusiones
Las bacterias multirresistentes representan un problema de gran actualidad e importancia. Esta revisión pone de manifiesto la existencia de un problema global provocado por la resistencia bacteriana. En concreto, se destacan las carbapenemasas, que es solo uno de los muchos mecanismos de resistencia desarrollados por bacterias. Nuestra revisión sugiere que, gran cantidad de las infecciones bacterianas que por ahora se pueden combatir de forma eficaz con antibióticos, según las evidencias, podrían ir en camino de convertirse en una amenaza contra la que la sociedad se encuentre indefensa y a un ritmo mucho mayor del que se podría presuponer. Por ello, es importante que se tome conciencia y se colabore a nivel global, haciendo un uso responsable de los antibióticos, tanto en el ámbito personal como en el industrial. Además, es imprescindible aumentar las inversiones en investigación, ya que han demostrado ser la principal vía para afrontar este y otros muchos retos sanitarios.
Declaraciones
Agradecimientos
Queremos agradecer al curso “Producción y traducción de artículos biomédicos” por darnos la oportunidad de llevar a cabo este proyecto. A todo su equipo, y en especial a Mario Rivera Izquierdo, Pablo Redruello Guerrero y Manuel González Alcaide por enseñarnos todo lo necesario para iniciarnos en el mundo de la investigación y publicación científica.
Conflinctos de interés
Los autores declaran no tener ningún conflicto de interés.
Financiación
Ninguna
Referencias
1. La OMS publica la lista de las bacterias para las que se necesitan urgentemente nuevos antibióticos. Disponible en: https://www.who.int/es/news/item/27-02-2017-who-publishes-list-of-bacteria-for-which-new-antibiotics-are-urgently-needed (acceso el 3 de abril de 2022).
2. Homepage | European Centre for Disease Prevention and Control Disponible en: https://www.ecdc.europa.eu/en (acceso el 3 de abril de 2022).
3. Sánchez-López E, Gomes D, Esteruelas G, Bonilla L, Lopez-Machado AL, Galindo R, et al. Metal-Based Nanoparticles as Antimicrobial Agents: An Overview. Nanomaterials. 2020;10(2):292.
4. Opal, Steven M.; Pop-Vicas, Aurora. Mecanismos moleculares de resistencia antimicrobiana en las bacterias. En: Bennett, John E., MD; Dolin, Raphael, MD; Blaser, Martin J., MD, editores. Mandell, Douglas y Bennett. Enfermedades infecciosas. Principios y práctica. Elsevier; 2020. 222-239.
5. Tenover FC. Mechanisms of antimicrobial resistance in bacteria. Am J Infect Control. 2006;34:S3-10; discussion S64-73.
6. Guía-ABE. Disponible en: https://www.guia-abe.es/ (acceso el 3 de abril de 2022).
7. Suárez C, Gudiol F. Antibióticos betalactámicos. Enfermedades Infecc Microbiol Clínica. 2009;27(2):116-29.
8. Calvo J, Martínez-Martínez L. Mecanismos de acción de los antimicrobianos. Enfermedades Infecc Microbiol Clínica. 2009;27(1):44-52.
9. Abushaheen MA, Muzaheed, Fatani AJ, Alosaimi M, Mansy W, George M, et al. Antimicrobial resistance, mechanisms and its clinical significance. Dis–Mon DM. 2020;66(6):100971.
10. Christaki E, Marcou M, Tofarides A. Antimicrobial Resistance in Bacteria: Mechanisms, Evolution, and Persistence. J Mol Evol. 2020;88(1):26-40.
11. Amaral L, Martins A, Spengler G, Molnar J. Efflux pumps of Gram-negative bacteria: what they do, how they do it, with what and how to deal with them. Front Pharmacol. 2014;4:168.
12. Rosolli GM, Arena F, Giani T. Mechanisms of Antibacterial Resistance. En: Jonathan Cohen, William G. Powderly, Steven M. Opal, editores. Infectious Diseases. 4th edition. Amsterdam: Elsevier; 2017. 1181-1196.
13. Ambler RP. Clasificación AMBLER. Philos Trans R Soc Lond B Biol Sci. 1980;289(1036):321-31.
14. Naas T, Dortet L, Iorga BI. Structural and Functional Aspects of Class A Carbapenemases. Curr Drug Targets. 2016;17(9):1006-28.
15. Queenan AM, Bush K. Carbapenemases: the Versatile β-Lactamases. Clin Microbiol Rev. 2007;20(3):440-58.
16. Rivera-Izquierdo M, Láinez-Ramos-Bossini AJ, Rivera-Izquierdo C, López-Gómez J, Fernández-Martínez NF, Redruello-Guerrero P, et al. OXA-48 Carbapenemase-Producing Enterobacterales in Spanish Hospitals: An Updated Comprehensive Review on a Rising Antimicrobial Resistance. Antibiot Basel Switz. 2021;10(1):89.
17. Nordmann P, Naas T, Poirel L. Global Spread of Carbapenemase-producing Enterobacteriaceae. Emerg Infect Dis. 2011;17(10):1791-89.
18. Pana ZD, Zaoutis T. Treatment of extended-spectrum β-lactamase-producing Enterobacteriaceae (ESBLs) infections: what have we learned until now? F1000Research. 2018;7:F1000 Faculty Rev-1347.
19. van Duin D, Doi Y. The global epidemiology of carbapenemase-producing Enterobacteriaceae. Virulence. 2016;8(4):460-9.
20. Souli M, Galani I, Antoniadou A, Papadomichelakis E, Poulakou G, Panagea T, et al. An Outbreak of Infection due to β-Lactamase Klebsiella pneumoniae Carbapenemase 2–Producing K. pneumoniae in a Greek University Hospital: Molecular Characterization, Epidemiology, and Outcomes. Clin Infect Dis. 2010;50(3):364-73.
21. Giakoupi P, Maltezou H, Polemis M, Pappa O, Saroglou G, Vatopoulos A, et al. KPC-2-producing Klebsiella pneumoniae infections in Greek hospitals are mainly due to a hyperepidemic clone. Euro Surveill Bull Eur Sur Mal Transm Eur Commun Dis Bull. 2009;14(21):19218.
22. Logan LK, Weinstein RA. The Epidemiology of Carbapenem-Resistant Enterobacteriaceae: The Impact and Evolution of a Global Menace. J Infect Dis. 2017;215:S28-36.
23. Rhomberg PR, Deshpande LM, Kirby JT, Jones RN. Activity of meropenem as serine carbapenemases evolve in US Medical Centers: monitoring report from the MYSTIC Program (2006). Diagn Microbiol Infect Dis. 2007;59(4):425-32.
24. Kaiser RM, Castanheira M, Jones RN, Tenover F, Lynfield R. Trends in Klebsiella pneumoniae carbapenemase-positive K. pneumoniae in US hospitals: report from the 2007-2009 SENTRY Antimicrobial Surveillance Program. Diagn Microbiol Infect Dis. 2013;76(3):356-60.
25. Logan LK, Renschler JP, Gandra S, Weinstein RA, Laxminarayan R. Carbapenem-Resistant Enterobacteriaceae in Children, United States, 1999–2012. Emerg Infect Dis. 2015;21(11):2014-21.
26. Nordmann P, Cuzon G, Naas T. The real threat of Klebsiella pneumoniae carbapenemase-producing bacteria. Lancet Infect Dis. 2009;9(4):228-36.
27. Chen L, Mathema B, Chavda KD, DeLeo FR, Bonomo RA, Kreiswirth BN. Carbapenemase-producing Klebsiella pneumoniae: molecular and genetic decoding. Trends Microbiol. 2014;22(12):686-96.
28. Martin RM, Bachman MA. Colonization, Infection, and the Accessory Genome of Klebsiella pneumoniae. Front Cell Infect Microbiol. 2018;8:4.
29. Wu W, Feng Y, Tang G, Qiao F, McNally A, Zong Z. NDM Metallo-β-Lactamases and Their Bacterial Producers in Health Care Settings. Clin Microbiol Rev. 2019;32(2):e00115-18.
30. Xiang T, Chen C, Wen J, Liu Y, Zhang Q, Cheng N, et al. Resistance of Klebsiella pneumoniae Strains Carrying blaNDM–1 Gene and the Genetic Environment of blaNDM–1. Front Microbiol. 2020;11:700.
31. Xu L, Sun X, Ma X. Systematic review and meta-analysis of mortality of patients infected with carbapenem-resistant Klebsiella pneumoniae. Ann Clin Microbiol Antimicrob. 2017;16:18.
32. Mairi A, Pantel A, Sotto A, Lavigne J-P, Touati A. OXA-48-like carbapenemases producing Enterobacteriaceae in different niches. Eur J Clin Microbiol Infect Dis. 2018;37(4):587-604.
33. Argente M, Miró E, Martí C, Vilamala A, Alonso-Tarrés C, Ballester F, et al. Molecular characterization of OXA-48 carbapenemase-producing Klebsiella pneumoniae strains after a carbapenem resistance increase in Catalonia. Enfermedades Infecc Microbiol Clin Engl Ed. 2019;37(2):82-8.
34. PRAN. Disponible en: https://resistenciaantibioticos.es/es (acceso el 3 de abril de 2022).
35. AEMPS. Disponible en: https://www.aemps.gob.es/ (acceso el 3 de abril de 2022).
36. León-Sampedro R, DelaFuente J, Díaz-Agero C, Crellen T, Musicha P, Rodríguez-Beltrán J, et al. Pervasive transmission of a carbapenem resistance plasmid in the gut microbiota of hospitalised patients. Nat Microbiol. 2021;6(5):606-16.
37. Aguilera-Alonso D, Escosa-García L, Goycochea-Valdivia WA, Soler-Palacín P, Saavedra-Lozano J, Rodrigo C, et al. [Position statement of the Spanish Association of Paediatrics-Spanish Society of Paediatric Infectious Diseases (AEP-SEIP) on the treatment of multidrug-resistant bacterial infections]. An Pediatr. 2019;91(5):351.e1-351.e13.
38. CNB – Centro Nacional de Biotecnología. Disponible en: http://www.cnb.csic.es/index.php/es/ (acceso el 3 de abril de 2022).
39. García-Fernández E, Koch G, Wagner RM, Fekete A, Stengel ST, Schneider J, et al. Membrane Microdomain Disassembly Inhibits MRSA Antibiotic Resistance. Cell. 2017;171(6):1354-1367.e20.
40. Hernando-Amado S, Laborda P, Valverde JR, Martínez JL. Rapid Decline of Ceftazidime Resistance in Antibiotic-Free and Sublethal Environments Is Contingent on Genetic Background. Mol Biol Evol. 2022;39(3):msac049.
41. Collateral sensitivity associated with antibiotic resistance plasmids | eLife. Disponible en: https://elifesciences.org/articles/65130#fig1s2 (acceso el 3 de abril de 2022).
42. López-Montesinos I, Horcajada JP. Oral and intravenous fosfomycin in complicated urinary tract infections. Rev Espanola Quimioter Publicacion Of Soc Espanola Quimioter. 2019;32 Suppl 1:37-44.
43. Epine. Disponible en: https://epine.es/ (acceso el 3 de abril de 2022).
AMU 2022. Volumen 4, Número 1
Fecha de envío: Fecha de aceptación: Fecha de publicación:
23/03/2022 04/04/2022 31/05/2022
Cita el artículo: Lirola-Andreu Laura, Ávila-Jiménez Ángel Francisco, Fernández-Mariscal Marta Andrea, Reinoso-Espín Ángela, Martínez-Martínez Silvia. La resistencia bacteriana. Generalidades, carbapenemasas y actualidad: una revisión narrativa. AMU. 2022; 4(1): 65-74